---
title: "Covid-19 Italy"
output:
flexdashboard::flex_dashboard:
orientation: rows
vertical_layout: fill
social: menu
source_code: embed
---
```{r setup, include=FALSE}
library(flexdashboard)
library(ggplot2)
library(highcharter)
library(gtrendsR)
library(lubridate)
library(dplyr)
library(stringr)
library(viridisLite)
library(DT)
library(magrittr)
library(dygraphs)
library(plotly)
library(xts)
library(viridis)
library(leaflet)
library(tmap)
```
```{r}
thm <-
hc_theme(
colors = c("#1a6ecc", "#434348", "#90ed7d"),
chart = list(
backgroundColor = "transparent",
style = list(fontFamily = "Source Sans Pro")),
xAxis = list( gridLineWidth = 1)
)
confirmed_color <- "purple"
active_color <- "#1f77b4"
recovered_color <- "forestgreen"
death_color <- "red"
```
```{r}
# covid x regioni
covid_italy <- read.csv("https://raw.githubusercontent.com/pcm-dpc/COVID-19/master/dati-regioni/dpc-covid19-ita-regioni.csv")
# covid nazionale
covid_italy_sum <- read.csv("https://raw.githubusercontent.com/pcm-dpc/COVID-19/master/dati-andamento-nazionale/dpc-covid19-ita-andamento-nazionale.csv")
# covid x province
covid_itpro <- read.csv("https://raw.githubusercontent.com/pcm-dpc/COVID-19/master/dati-province/dpc-covid19-ita-province.csv")
```
```{r}
covid_italy %<>%
mutate(data = date(data)) %>%
mutate(regione = as.character(denominazione_regione)) %>%
select(-denominazione_regione,
-stato,
-codice_regione,
-lat,
-long,
-note_it,
-note_en) %>%
mutate(regione = ifelse(.$regione == "Trento","Trentino-Alto Adige",.$regione))%>%
mutate( regione = ifelse(.$regione == "Bolzano","Trentino-Alto Adige",.$regione)) %>%
mutate(regione = ifelse(.$regione == "P.A. Trento","Trentino-Alto Adige",.$regione))%>%
mutate( regione = ifelse(.$regione == "P.A. Bolzano","Trentino-Alto Adige",.$regione)) %>%
group_by(regione,data) %>%
summarize_all(~sum(.)) %>%
ungroup(regione) %>%
arrange(desc(data),desc(totale_casi))
```
```{r}
covid_italy_sum %<>%
mutate(data = date(data))
```
```{r}
today <- max(covid_italy$data)
covid_italy_map <-
covid_italy %>%
filter(data == today) %>%
select("regione","totale_positivi")
```
```{r}
map_ita <- readRDS("map_ita_json.RDS")
region_list <- c("Piemonte","Valle d'Aosta","Lombardia",
"Trentino-Alto Adige","Veneto",
"Friuli Venezia Giulia","Liguria","Emilia-Romagna",
"Toscana","Umbria","Marche","Lazio",
"Abruzzo","Molise","Campania","Puglia","Basilicata",
"Calabria","Sicilia","Sardegna")
for(i in 1:20){
map_ita[["features"]][[i]][["properties"]][["name"]] <- region_list[i]
# cat(map_ita[["features"]][[i]][["properties"]][["name"]],"\n")
}
```
```{r}
covid_itpro %<>%
mutate (data=ymd_hms(data))%>%
mutate(data = date(data))
geo_italy <- geojsonio::geojson_read("limits_IT_provinces.geojson", what = "sp")
pal <- colorNumeric("viridis", NULL)
geo_italy$prov_name <- as.character(geo_italy$prov_name)
geo_italy$prov_name <- ifelse(geo_italy$prov_name == "Valle d'Aosta/Vallée d'Aoste","Aosta",geo_italy$prov_name )
geo_italy$prov_name <- ifelse(geo_italy$prov_name == "Bolzano/Bozen","Bolzano",geo_italy$prov_name )
geo_italy$prov_name <- ifelse(geo_italy$prov_name == "Massa-Carrara","Massa Carrara",geo_italy$prov_name )
covid_itpro %<>%
filter(denominazione_provincia != "In fase di definizione/aggiornamento") %>%
filter(data == today) %>%
mutate(denominazione_provincia = as.character(denominazione_provincia)) %>%
mutate(denominazione_provincia =
ifelse( .$denominazione_provincia=="Forlì-Cesena",
"Forlì-Cesena",.$denominazione_provincia))
covid_province <-
sp::merge(geo_italy,covid_itpro,by.x="prov_name", by.y="denominazione_provincia")
```
Sommario
=======================================================================
Row {data-height=200}
-------------------------------------
### confirmed {.value-box}
```{r}
tot <- covid_italy_sum %>%
filter(data == today) %>%
select(totale_casi ) %>%
pull
tot_1 <- covid_italy_sum %>%
filter(data == today-1) %>%
select(totale_casi ) %>%
pull
valueBox(value = paste(tot, "", " (+",
round(100 * (tot-tot_1)/tot_1 , 1),
"%)",
sep = ""),
caption = "Casi Totali",
icon = "fas fa-user-md",
color = confirmed_color)
```
### active {.value-box}
```{r}
pos <-
covid_italy_sum %>%
filter(data == today) %>%
select(totale_positivi) %>%
pull
pos_1 <-
covid_italy_sum %>%
filter(data == today -1) %>%
select(totale_positivi) %>%
pull
valueBox(value = paste(as.character(pos), " (+",
round(100 * (pos-pos_1)/pos_1 , 1),
"%)", sep = ""),
caption = "Attualmente Positivi", icon = "fas fa-ambulance",
color = active_color)
```
### recovered {.value-box}
```{r}
rec <-
covid_italy_sum %>%
filter(data == today) %>%
select(dimessi_guariti ) %>%
pull
rec_1 <-
covid_italy_sum %>%
filter(data == today -1) %>%
select(dimessi_guariti ) %>%
pull
valueBox(value = paste(format(rec, big.mark = ","), " (+",
round(100 * (rec-rec_1) / rec_1, 1),
"%)", sep = ""),
caption = "Guariti", icon = "fas fa-heartbeat",
color = recovered_color)
```
### death {.value-box}
```{r}
death <-
covid_italy_sum %>%
filter(data == today) %>%
select(deceduti) %>%
pull
death_1 <-
covid_italy_sum %>%
filter(data == today -1) %>%
select(deceduti) %>%
pull
valueBox(value = paste(death, " (+",
round(100 * (death-death_1) / death_1, 1),
"%)", sep = ""),
caption = "Deceduti",
icon = "fas fa-heart-broken",
color = death_color)
```
Row {data-height=1000}
-----------------------------------------------------------------------
### Mappa Contagi
```{r}
n <- 4
stops <- data.frame(q = 0:n/n,
c = substring(viridis(n + 1), 0, 7)[(n+1):1],
stringsAsFactors = FALSE)
stops <- list_parse2(stops)
highchart(type = "map") %>%
hc_add_series_map(map = map_ita,
df = covid_italy_map,
joinBy = c("name","regione"),
value = "totale_positivi",
name="Totale Positivi") %>%
hc_colorAxis(stops= stops,min=min(covid_italy_map$totale_positivi)) %>%
hc_legend(valueDecimals = 0, valueSuffix = "%") %>%
hc_mapNavigation(enabled = TRUE) %>%
hc_add_theme(thm)
```
### Cronologia contagi {data-width=700}
```{r}
positivi <- xts(covid_italy_sum$totale_positivi, covid_italy_sum$data)
deceduti <- xts(covid_italy_sum$deceduti, covid_italy_sum$data)
dimessi <- xts(covid_italy_sum$dimessi_guariti, covid_italy_sum$data)
totali <- xts(covid_italy_sum$totale_casi,covid_italy_sum$data)
covid_italy_time <- merge.xts(totali,positivi,dimessi,deceduti)
```
```{r}
dygraph(covid_italy_time) %>%
dyRangeSelector()
```
Analisi
=======================================================================
### Mappa contagi
```{r}
breaks = c(0, 100, 500, 1000, 2500, 5000, 10000)
map_ita = tm_shape(covid_province) +
tm_polygons(col="totale_casi",breaks=breaks,
popup.vars=c("totale casi:"="totale_casi"))
tmap_leaflet(map_ita)
```
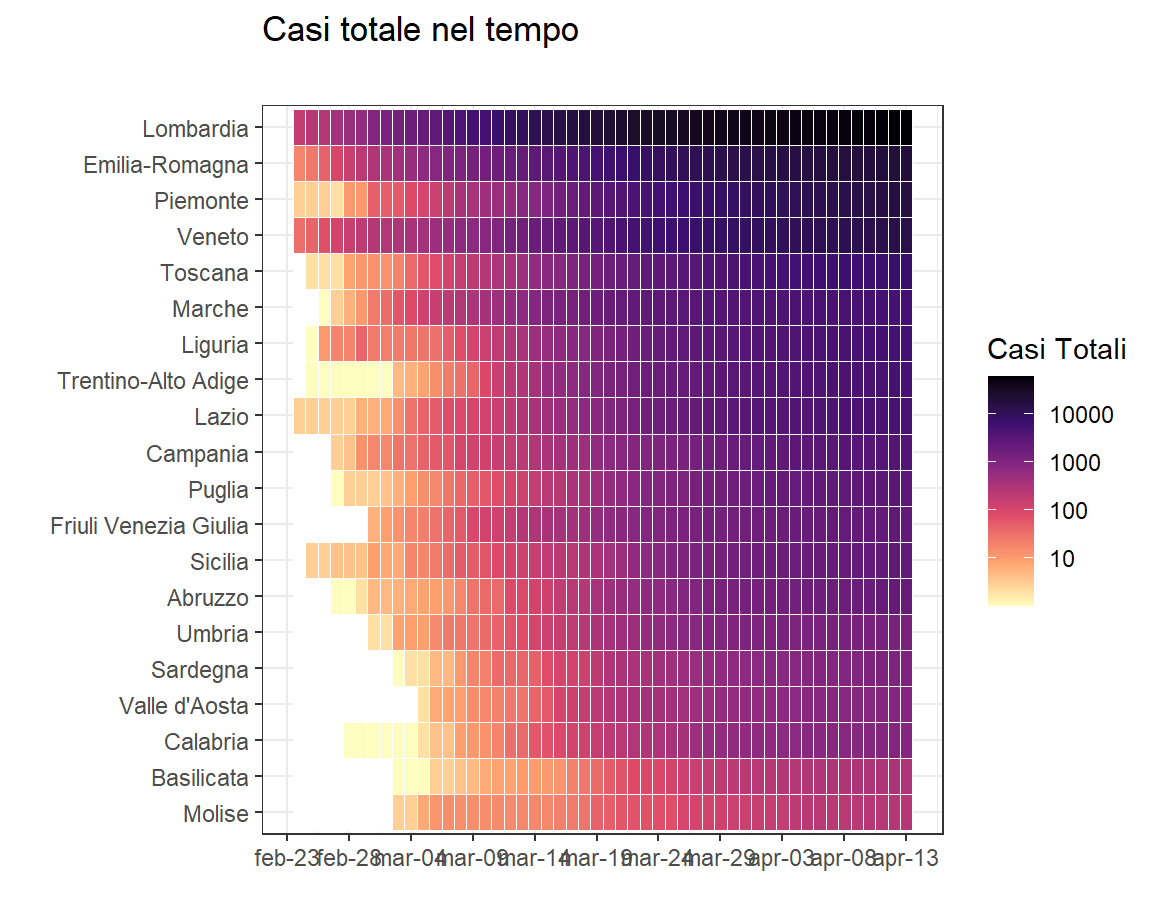
### Heatmap
```{r}
# https://gist.github.com/jebyrnes/b05f4ca12a1f791030eb591720d6c70e
cmax <- log10(max(covid_italy$totale_casi))/5
lab_e <- round(10^c(0:5*cmax),0)
lab_txt <- paste0(covid_italy$regione, ": ", covid_italy$totale_casi)
#gp <-
ggplot(covid_italy,aes(x = data, y = reorder(regione, totale_casi),
fill = log10(totale_casi),
text= lab_txt)) +
geom_tile(size = 0.2, colour="white") +
scale_fill_viridis_c(option = "A", na.value="white", direction=-1,
breaks = 1:4,
labels = c(10,100,1000,10000),
guide = guide_colorbar("Casi Totali")) +
scale_x_date(date_breaks = "5 days" , date_labels = "%b-%d")+
xlab("") + ylab("") +
ggtitle("Casi totale nel tempo", subtitle = "") +
theme_bw()
#ggplotly(gp, tooltip = "text")
```
Download Data
=======================================================================
Row {data-height=1200}
-------------------------------------
```{r}
datatable(
covid_italy,
extensions = 'Buttons',
options = list(
pageLength = 24,
dom = 'Bfrtip',
buttons = c('copy', 'csv', 'excel', 'pdf', 'print')
))
```
About
=======================================================================
**Dashboard sul Coronavirus in Italia**
Questa dashboard sul Coronavirus offre una panoramica dell'epidemia di Coronavirus COVID-19 in Italia e analizza i dati ufficiale che fornisce la [Protezione Civile nella propria repository](https://github.com/pcm-dpc/COVID-19)
La dashboard sarà aggiornata verso le 20:00 di ogni sera dopo che i dati saranno rilasciati.
Il codice può essere reperito presso la repository di github al seguente [link](https://github.com/alessiopassalacqua/covid19_italy)
by Alessio Passalacqua
alessio.passalacqua@gmail.com